今日から写経する本を紹介します
新しい本
今日からはこちらの Pythonで体験するベイズ推論 PyMCによるMCMC入門
を写経していくことにしました。
変更理由
理由は二つあります。
一つは、今まで読んでいた 世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
に飽きたことです。主に写経したのは中盤のあたりでしたが、それ以降の21章以降からは統計の読み物と機械学習の簡単な導入が書かれているような印象でした。 機械学習のコードを書くなら、他のもっと詳しい本で勉強したいと思ったので、21以降の章は写経しないことにしました。
理由の二つ目は、ベイズ理論へのざっくりとした導入を勉強したかったからです。僕は、数か月前まで、パターン認識と機械学習 上とパターン認識と機械学習 下 (ベイズ理論による統計的予測)
を読んでいましたが、説明がなかなか読み取りにくく理解できない部分も多い本でした。ベイズ理論を軸に議論している本でしたが、ベイズ理論の説明が乏しく、未だにベイズ理論が何なのかが理解できていません。
どんな本なの?
そこでPythonで体験するベイズ推論 PyMCによるMCMC入門を読むことにしました。初めの数ページを読んだ限り、文章が非常に丁寧な印象があり、書いてあることが理解しやすかったです。内容的にも、PyMCというライブラリの使い方の解説が多そうで、今の僕にはない知識を得ることができそうでした。
というわけで、本日からPythonで体験するベイズ推論 PyMCによるMCMC入門をがしがし写経していきます。
Python言語によるプログラミングイントロダクション 第2版 写経(20章)
世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
の写経をしたので記録を公開しておきます。
Jupyter Labで実行したものをペーストしているので、ちょっとした正誤表になるかもしれません。
この記事では、20章をまとめておきます。
写経
import random import pylab
pylab.rc('figure', dpi=200)
20.3 ベイズ更新
code 20.1
def calc_bays(prior_A, prob_b_if_A, prob_B): return prior_A*prob_b_if_A/prob_B prior_A = 1/3 prob_6_if_A = 1/5 prob_6 = (1/5 + 1/6 + 1/7)/3 post_A = calc_bays(prior_A, prob_6_if_A, prob_6) print(f'タイプAである確率 = {round(post_A, 4)}') post_A = calc_bays(post_A, prob_6_if_A, prob_6) print(f'タイプAである確率 = {round(post_A, 4)}')
タイプAである確率 = 0.3925
タイプAである確率 = 0.4622
post_A = calc_bays(prior_A, 1 - prob_6_if_A, 1 - prob_6) print(f'タイプAである確率 = {round(post_A, 4)}') post_A = calc_bays(post_A, 1 - prob_6_if_A, 1 - prob_6) print(f'タイプAである確率 = {round(post_A, 4)}')
タイプAである確率 = 0.3212
タイプAである確率 = 0.3096
prior_A = 9/10 post_A = calc_bays(prior_A, 1 - prob_6_if_A, 1 - prob_6) print(f'タイプAである確率 = {round(post_A, 4)}') post_A = calc_bays(post_A, 1 - prob_6_if_A, 1 - prob_6) print(f'タイプAである確率 = {round(post_A, 4)}')
タイプAである確率 = 0.8673
タイプAである確率 = 0.8358
code 20.2
def bays_update(num_rolls=200, prior_A=9/10): post_A = prior_A for i in range(num_rolls + 1): if i%(num_rolls//10) == 0: print(f'{i}回振った時点でタイプAである確率 = {round(post_A, 4)}') is_six = random.random() <= 1/7 if is_six: post_A = calc_bays(post_A, prob_6_if_A, prob_6) else: post_A = calc_bays(post_A, 1 - prob_6_if_A, 1 - prob_6)
bays_update()
0回振った時点でタイプAである確率 = 0.9
20回振った時点でタイプAである確率 = 0.7834
40回振った時点でタイプAである確率 = 0.4567
60回振った時点でタイプAである確率 = 0.5936
80回振った時点でタイプAである確率 = 0.6314
100回振った時点でタイプAである確率 = 1.0028
120回振った時点でタイプAである確率 = 1.0667
140回振った時点でタイプAである確率 = 0.6218
160回振った時点でタイプAである確率 = 0.6614
180回振った時点でタイプAである確率 = 0.7035
200回振った時点でタイプAである確率 = 0.7483
メモ
どうやら、事後確率が1以上になることがあるようだ。というか、そもそも
print(f'prob_6_if_A/prob_6 = {prob_6_if_A/prob_6}')
prob_6_if_A/prob_6 = 1.177570093457944
なので、random.random() <= 1/7でTrueが続くと事後確率が1以上になるのは当たり前だ。確立を論じている章としていかがなものか。
次の章読んでこういうコードが続くならこの本は切ろうかな・・・
メモ
ベイズ更新の様子を可視化するためにプロットを行ってみた。上でも述べたが、このベイズ更新の式は確率論的に誤りがあるので、参考程度にプロットしたまでである。
def bays_update(num_rolls=200, prior_A=9/10): prob_6_if_A = 1/5 prob_6 = (1/5 + 1/6 + 1/7)/3 post_A = prior_A post_As = [] post_As.append(post_A) for i in range(num_rolls + 1): is_six = random.random() <= 1/7 if is_six: post_A = calc_bays(post_A, prob_6_if_A, prob_6) post_As.append(post_A) else: post_A = calc_bays(post_A, 1 - prob_6_if_A, 1 - prob_6) post_As.append(post_A) return post_As def plot_bays_update(num_rolls=200, prior_A=9/10, num_trials=10): for i in range(1, 1 + num_trials): post_As = bays_update(num_rolls, prior_A) pylab.plot(post_As, label=f'trial {i}') pylab.title('Bays uppdate') pylab.xlabel('Dice throw') pylab.ylabel('Post probability') pylab.legend(loc='best')
plot_bays_update(prior_A=1/3)


世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
- 作者: ジョン・V.グッターグ,John V. Guttag,久保幹雄
- 出版社/メーカー: 近代科学社
- 発売日: 2017/09/01
- メディア: 単行本
- この商品を含むブログを見る
Python言語によるプログラミングイントロダクション 第2版 写経(19章)
世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
の写経をしたので記録を公開しておきます。
Jupyter Labで実行したものをペーストしているので、ちょっとした正誤表になるかもしれません。
この記事では、19章をまとめておきます。
写経
この章から関数と変数の名前をpep8に従うように変更してある。
以下使用モジュールとrcパラメータの設定。
import pylab import scipy as sp import scipy.stats import random import statistics as stat import collections
pylab.rc('figure', dpi=200)
19.1 有意性のチェック
code 19.1
def plot_tdist(t_stat=-2.13165598142, num_bins=1000, num_samples=10000000, deg_of_freedom=198): t_dist = [sp.random.standard_t(deg_of_freedom) for i in range(num_samples)] pylab.hist(t_dist, bins=num_bins, weights=num_samples*[1.0/num_samples]) pylab.axvline(t_stat, color='w') pylab.axvline(-t_stat, color='w') pylab.title(f'T-distribution with {deg_of_freedom} Degrees of Freedom') pylab.xlabel('T-statistic') pylab.ylabel('Probability')
plot_tdist()

code 19.2
メモ
controlTimesとtreatmentTimesが急に出てきたけど、ファイルなどから読み込まれてはいないから使えない。なので、19.pyを参考にして生成した。
後で気が付いたが、code 19.3にcontrolTimesとtreatmentTimesを生成するコードが書いてあった。ふざけんな
random.seed(0) treatment_times = [random.gauss(119.5, 5.0) for i in range(100)] control_times = [random.gauss(120, 4.0) for i in range(100)]
def calc_tstat_pval(): treatment_mean = stat.mean(treatment_times) control_mean = stat.mean(control_times) print(f'Treatment mean - control mean ={treatment_mean - control_mean} minutes') two_sample_test = sp.stats.ttest_ind(treatment_times, control_times, equal_var=False) print(f'The t-statistic from two-sample test is {two_sample_test[0]}') print(f'The p-value from two-sample test is {two_sample_test[1]}')
calc_tstat_pval()
Treatment mean - control mean =-0.9626159148482003 minutes
The t-statistic from two-sample test is -1.4677518436370114
The p-value from two-sample test is 0.1439256440649787
19.2 p値に注意せよ
メモ
303ページに別の標本でp値を計算していたので、僕もやってみた。random.seed(1)を実行することで、別の乱数から生成された標本が得られるの。(別に、random.seedを使わなくても異なる乱数は得られるのだが、seedの理解の為にやった。)
random.seed(1) treatment_times = [random.gauss(119.5, 5.0) for i in range(100)] control_times = [random.gauss(120, 4.0) for i in range(100)]
def calc_tstat_pval(): treatment_mean = stat.mean(treatment_times) control_mean = stat.mean(control_times) print(f'Treatment mean - control mean ={treatment_mean - control_mean} minutes') two_sample_test = sp.stats.ttest_ind(treatment_times, control_times, equal_var=False) print(f'The t-statistic from two-sample test is {two_sample_test[0]}') print(f'The p-value from two-sample test is {two_sample_test[1]}')
calc_tstat_pval()
Treatment mean - control mean =-1.4022836116031243 minutes
The t-statistic from two-sample test is -2.277359505985212
The p-value from two-sample test is 0.023866362341807106
figure 19.4
def plot_pvals(num_trial=10000, bins=100): pvals = [] pval_small = 0 for i in range(num_trial): treatment_times = [random.gauss(119.5, 5.0) for i in range(100)] control_times = [random.gauss(120, 4.0) for i in range(100)] pval = sp.stats.ttest_ind(treatment_times, control_times, equal_var=False)[1] pvals.append(pval) if pval < 0.05: pval_small += 1 pval_median = stat.median(pvals) pval_mean = stat.mean(pvals) fraction_small = pval_small/num_trial pylab.hist(pvals, bins=bins, weights=num_trial*[1/num_trial]) pylab.title('P-values from Actual Distribution') pylab.xlabel('p-value') pylab.ylabel('Probability') pylab.annotate(f'Mean = {round(pval_mean, 4)}, Median = {round(pval_median, 4)}\nFraction below 0.05 = {round(fraction_small, 4)}', xy=(0.1, 0.7), xycoords='axes fraction')
plot_pvals()

1標本検定追加
#random.seed(1) treatment_times = [random.gauss(119.5, 5.0) for i in range(100)] control_times = [random.gauss(120, 4.0) for i in range(100)] def calc_tstat_pval(): treatment_mean = stat.mean(treatment_times) control_mean = stat.mean(control_times) print(f'Treatment mean - control mean ={treatment_mean - control_mean} minutes') two_sample_test = sp.stats.ttest_ind(treatment_times, control_times, equal_var=False) print(f'The t-statistic from two-sample test is {two_sample_test[0]}') print(f'The p-value from two-sample test is {two_sample_test[1]}') one_sample_test = sp.stats.ttest_1samp(treatment_times, 120) print(f'The t-statistic from one-sample test is {one_sample_test[0]}') print(f'The p-value from one-sample test is {one_sample_test[1]}') calc_tstat_pval()
Treatment mean - control mean =-0.3566556416159159 minutes
The t-statistic from two-sample test is -0.5549758199376972
The p-value from two-sample test is 0.5795487828148798
The t-statistic from one-sample test is -1.5022019326317575
The p-value from one-sample test is 0.13622803414860576
19.4 有意性の有無
def john1(num_games=1273, lyndsay_wins = 666): outcomes = lyndsay_wins*[1.0] + (num_games - lyndsay_wins)*[0.0] print(f'The p-value from a one-sample test is {sp.stats.ttest_1samp(outcomes, 0.5)[1]}')
john1()
The p-value from a one-sample test is 0.0982205871243577
code 19.4
def lyndsay(num_games=1273, lyndsay_wins=666, num_trials=1000): at_least = 0 for t in range(num_trials): l_wins = 0 for g in range(num_games): if random.random() < 0.5: l_wins += 1 if l_wins >= lyndsay_wins: at_least += 1 print(f'Probability of result at least this extreame by accident = {at_least/num_trials}')
lyndsay()
Probability of result at least this extreame by accident = 0.046
code 19.5
def john2(num_games=1273, lyndsay_wins=666, num_trials=1000): at_least = 0 for t in range(num_trials): l_wins, j_wins = 0, 0 for g in range(num_games): if random.random() < 0.5: l_wins += 1 else: j_wins += 1 if l_wins >= lyndsay_wins or j_wins >= lyndsay_wins: at_least += 1 print(f'Probability of result at least this extreame by accident = {at_least/num_trials}')
john2()
Probability of result at least this extreame by accident = 0.088
figure 19.5
def plot_fig_19_5(mean_diff=0.5, stdev=5, num_trials=1000, start=1, stop=3000, step=50): mean1 = 0 mean2 = mean1 + mean_diff mean_pvals=[] for i in range(start, stop, step): pvals = [] for t in range(num_trials): pvals.append(sp.stats.ttest_ind([random.gauss(mean1, stdev) for j in range(i)], [random.gauss(mean2, stdev) for j in range(i)], equal_var=False)[1]) mean_pvals.append(stat.mean(pvals)) pylab.title(f'Gaussians with SD={stdev}, Means of {mean1} & {mean2}') pylab.plot(range(start, stop, step), mean_pvals, label='Mean p-value') pylab.xlabel('Sample size') pylab.ylabel('P-value') pylab.semilogy() pylab.axhline(0.05, linestyle='-', label='p = 0.05') pylab.axhline(0.01, linestyle='--', label='p = 0.01') pylab.legend(loc='best')
plot_fig_19_5()

19.6 多重仮説検定
bm_results2012_txt='bm_results2012.txt'
def get_BM_data(filename): data = {'name': [], 'gender': [], 'age':[], 'division': [], 'country': [], 'time': []} with open(filename) as f: for line in f.readlines(): split = line.split(',') data['name'].append(split[0]) data['gender'].append(split[1]) data['age'].append(split[2]) data['division'].append(split[3]) data['country'].append(split[4]) data['time'].append(float(split[5][:-1])) return data
data = get_BM_data(bm_results2012_txt)
code 19.6
countries_to_compare = ['BEL', 'BRA', 'FRA', 'JPN', 'ITA'] def fem_time_per_ctry(data, countries_to_compare): country_times = collections.defaultdict(list) for i in range(len(data['name'])): if data['country'][i] in countries_to_compare and data['gender'][i] == 'F': country_times[data['country'][i]].append(data['time'][i]) return country_times def comp_time(data, countries_to_compare): country_times = fem_time_per_ctry(data, countries_to_compare) for c1 in countries_to_compare: for c2 in countries_to_compare: if c1 < c2: pval = sp.stats.ttest_ind(country_times[c1], country_times[c2], equal_var=False)[1] if pval < 0.05: print(f'{c1} and {c2} have significantly different means, p-value = {round(pval, 4)}')
comp_time(data, countries_to_compare)
ITA and JPN have significantly different means, p-value = 0.025
code 19.7
メモ
このコードはどうやら、ガウス分布からサンプリングした値をpopulationリストに5000個保存して、populationリストからサンプリングしているようだ。どうせだったらガウス分布から直接サンプリングすればいいのにと思ったのでそうやるようなプログラムに書き直してみた。
あと、テキストのsampleSize=30はsampleSize=200の間違いなのではないだろうかと思う。
def mult_hyp(num_hyps=20, sample_size=200): sample = collections.defaultdict(list) for i in range(num_hyps): sample[1].append([random.gauss(0, 1) for i in range(sample_size)]) sample[2].append([random.gauss(0, 1) for i in range(sample_size)]) num_sig = 0 for i in range(num_hyps): if sp.stats.ttest_ind(sample[1][i], sample[2][i])[1] < 0.05: num_sig += 1 print(f'Number of statistically significant (p < 0.05) result = {num_sig}')
mult_hyp()
Number of statistically significant (p < 0.05) result = 0
メモ
mult_hyp関数の結果は1になったり0だったりと安定しないので、どんな感じの分布なのかを見てみるためのヒストグラムを書いてみた。
def mult_hyp(num_hyps=20, sample_size=200): sample = collections.defaultdict(list) for i in range(num_hyps): sample[1].append([random.gauss(0, 1) for i in range(sample_size)]) sample[2].append([random.gauss(0, 1) for i in range(sample_size)]) num_sig = 0 for i in range(num_hyps): if sp.stats.ttest_ind(sample[1][i], sample[2][i])[1] < 0.05: num_sig += 1 # print(f'Number of statistically significant (p < 0.05) result = {num_sig}') return num_sig def plot_num_sig(num_trial=1000, num_hyps=20, sample_size=200): num_sigs = [mult_hyp(num_hyps, sample_size) for i in range(num_trial)] min_sig = min(num_sigs) max_sig = max(num_sigs) bins = max_sig - min_sig + 1 pylab.hist(num_sigs, range=(min_sig - 0.5, max_sig + 0.5), density=True, bins=bins) pylab.title('Probability of significants') pylab.xlabel('Significants') pylab.ylabel('Probability')
plot_num_sig()

なるほど。稀に、20回のうちにp値が0.05を下回るとき6回あるときがあるのか。
そもそも、正規分布から200個サンプリングした二組のt-検定量がどんな感じなんだ?と思って、最後にプロットしてみた。
def plot_pval(num_trials=100000, sample_size=200, sigma=1): t_stats = [] for i in range(num_trials): sample = {} sample[1] = ([random.gauss(0, sigma) for i in range(sample_size)]) sample[2] = ([random.gauss(0, sigma) for i in range(sample_size)]) ttest = sp.stats.ttest_ind(sample[1], sample[2]) t_stats.append(ttest[0]) pylab.hist(t_stats, density=True, ) pylab.title('Histogram of t-statistics') pylab.ylabel('probability') pylab.xlabel('the t-statistic')
plot_pval()

どうやら、このt-検定の結果はだいたい正規分布に従うようだ。
おまけ
random.seedについて
random.seedの意味が分からなかったので少し遊んでみた。
ドキュメントはこれrandom.seed(a=None, version=2)。
ここを読む限り、乱数の生成にはOSの時刻が使われているようだが、random.seedを実行することで、生成される乱数を制御できるようだ。実際にrandom.seedを実行するのとしないのを比較したらその違いがよく分かった。random.seed(0)を実行しないと各乱数の生成で得られるリストは異なるものになるが、実行すると毎回同じ乱数が得られる。
#random.seed(0) [random.random() for i in range(4)]
[0.8159130965336595,
0.1006075202160962,
0.14635848891230385,
0.6976706401912388]
#random.seed(0) [random.random() for i in range(4)]
[0.5112747213686085,
0.4049341374504143,
0.7837985890347726,
0.30331272607892745]
random.seed(0) [random.random() for i in range(4)]
[0.8444218515250481,
0.7579544029403025,
0.420571580830845,
0.25891675029296335]
random.seed(0) [random.random() for i in range(4)]
[0.8444218515250481,
0.7579544029403025,
0.420571580830845,
0.25891675029296335]

世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
- 作者: ジョン・V.グッターグ,John V. Guttag,久保幹雄
- 出版社/メーカー: 近代科学社
- 発売日: 2017/09/01
- メディア: 単行本
- この商品を含むブログを見る
Python言語によるプログラミングイントロダクション 第2版 写経(18章)
世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
の写経をしたので記録を公開しておきます。 この記事では、17章をまとめておきます。
Jupyter Labで実行したものをペーストしているので、ちょっとした正誤表になるかもしれません。
以下、使用モジュールとrcParameterの設定。
import csv import pylab import collections import statistics as stat import math
pylab.rc('figure', dpi=200)
写経
18.1 バネの振舞い
springDataTxt = 'springData.txt'
code 18.1
def getData(fileName): distances = [] masses = [] with open(fileName, 'rt', newline='') as fin: csvReader = csv.reader(fin, delimiter=' ') # csvモジュールの練習で、ダイアレクトを設定してみた。 for line in csvReader: try: # ヘッダーはハンドリングする。float関数でValueErrorがでることを利用した。 distances.append(float(line[0])) masses.append(float(line[1])) except ValueError: continue return (masses, distances) # テキストファイルの中身並び順をわざわざ変えるの気持ち悪すぎる・・・
code 18.2
def plotData(inputFile): masses, distances = getData(inputFile) distances = pylab.array(distances) masses = pylab.array(masses) forces = masses*9.81 pylab.plot(forces, distances, 'bo', label = 'Measured displacements') pylab.title('Measured Displacement of Spring') pylab.xlabel('|Force| (Newtons)') pylab.ylabel('Distance (meters)')
plotData(springDataTxt)

18.1.1 線形回帰による適合
code 18.3
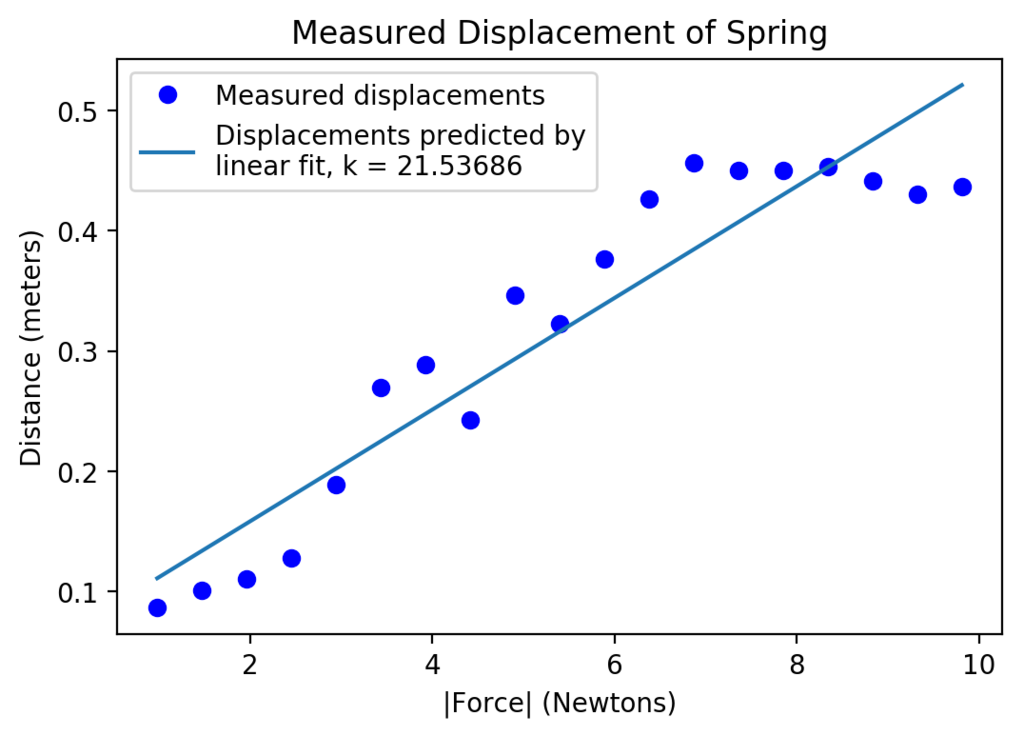
def fitData(inputFile): masses, distances = getData(inputFile) distances = pylab.array(distances) masses = pylab.array(masses) forces = masses*9.81 pylab.plot(forces, distances, 'bo', label = 'Measured displacements') pylab.title('Measured Displacement of Spring') pylab.xlabel('|Force| (Newtons)') pylab.ylabel('Distance (meters)') a, b = pylab.polyfit(forces, distances, 1) predictedDistances = a*forces + b k = 1.0/a pylab.plot(forces, predictedDistances, label=f'Displacements predicted by\nlinear fit, k = {round(k, 5)}') pylab.legend(loc='best')
fitData(springDataTxt)
code 18.3 (3次曲線追加)
def fitData(inputFile): masses, distances = getData(inputFile) distances = pylab.array(distances) masses = pylab.array(masses) forces = masses*9.81 pylab.plot(forces, distances, 'bo', label = 'Measured displacements') pylab.title('Measured Displacement of Spring') pylab.xlabel('|Force| (Newtons)') pylab.ylabel('Distance (meters)') a, b = pylab.polyfit(forces, distances, 1) predictedDistances = a*forces + b k = 1.0/a pylab.plot(forces, predictedDistances, label=f'Displacements predicted by\nlinear fit, k = {round(k, 5)}') fit = pylab.polyfit(forces, distances, 3) predictedDistances = pylab.polyval(fit, forces) pylab.plot(forces, predictedDistances, 'k:', label = 'cubic fit') pylab.legend(loc='best')
fitData(springDataTxt)

指練習 (オリジナル)
テキストの指練習の指示が曖昧過ぎる(同じ図をプロットするようにしろってなんだよ。x軸の最大値10のくせに図18.5は14以上までプロットしてるぞ・・・わからん・・)ので、各次数のプロットをやらせてみた。
データの数が19なので、十八次の曲線までプロットしてみよう。
def fitData(inputFile, start=1, stop=18, step=1): masses, distances = getData(inputFile) distances = pylab.array(distances) masses = pylab.array(masses) forces = masses*9.81 pylab.plot(forces, distances, 'bo', label = 'Measured displacements') pylab.title('Measured Displacement of Spring') pylab.xlabel('|Force| (Newtons)') pylab.ylabel('Distance (meters)') for i in range(start, stop + 1, step): fit = pylab.polyfit(forces, distances, i) predictedDistances = pylab.polyval(fit, forces) pylab.plot(forces, predictedDistances, label = f'digree: {i}') pylab.legend(loc='best')
fitData(springDataTxt, start=1, stop=9)

fitData(springDataTxt, start=10, stop=16)
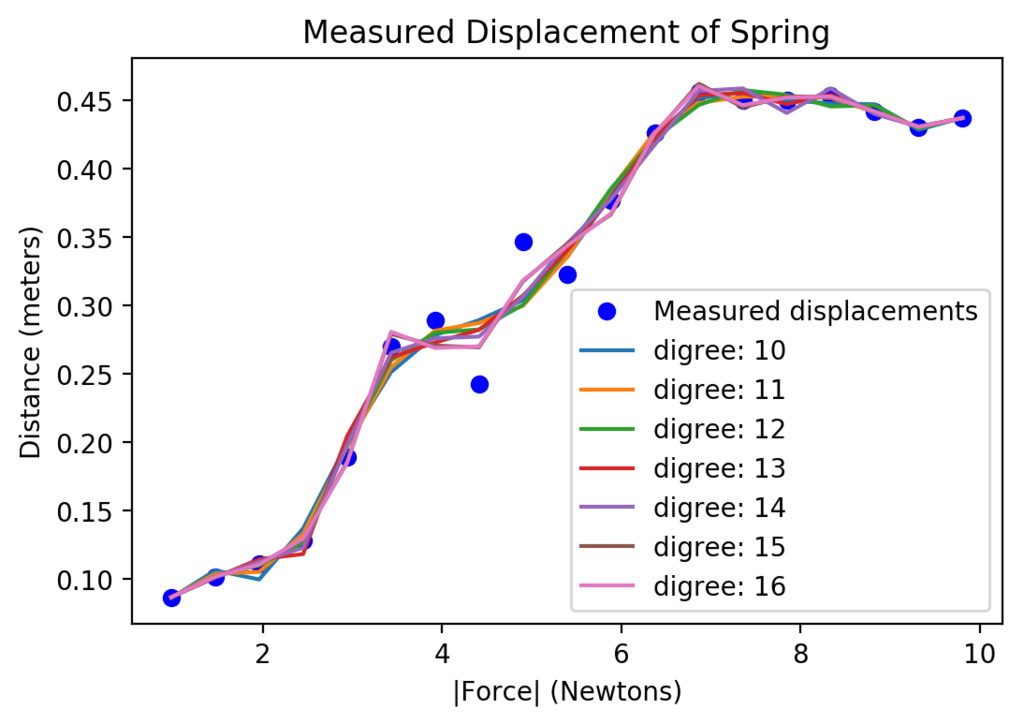
fitData(springDataTxt, start=17, stop=18)
C:\Users\ktsutsum\Anaconda3\lib\site-packages\ipykernel_launcher.py:11: RankWarning: Polyfit may be poorly conditioned
# This is added back by InteractiveShellApp.init_path()
C:\Users\ktsutsum\Anaconda3\lib\site-packages\ipykernel_launcher.py:11: RankWarning: Polyfit may be poorly conditioned
# This is added back by InteractiveShellApp.init_path()

十八次の多項式ならすべての点を通るようなグラフが欠けるかと予想してたけど、どうやら無理っぽい。Polyfit may be poorly conditionedってメッセージがでてるので、これらの点にはなんらかの線形関係があるのかな?
# This is added back by InteractiveShellApp.init_path()は意味わからんな。
同じようなメッセージは直線を四次でフィッティングさせようとしたときも出た。
fit = pylab.polyfit([1, 2, 3], [2, 4, 6], 4) predictedDistances = pylab.polyval(fit, [1, 2, 3]) pylab.plot([1, 2, 3], predictedDistances)
C:\Users\ktsutsum\Anaconda3\lib\site-packages\ipykernel_launcher.py:1: RankWarning: Polyfit may be poorly conditioned
"""Entry point for launching an IPython kernel.
[<matplotlib.lines.Line2D at 0x23e1d2e6e10>]

3点なら問題なくフィッティング出来た。これで満足しよう。
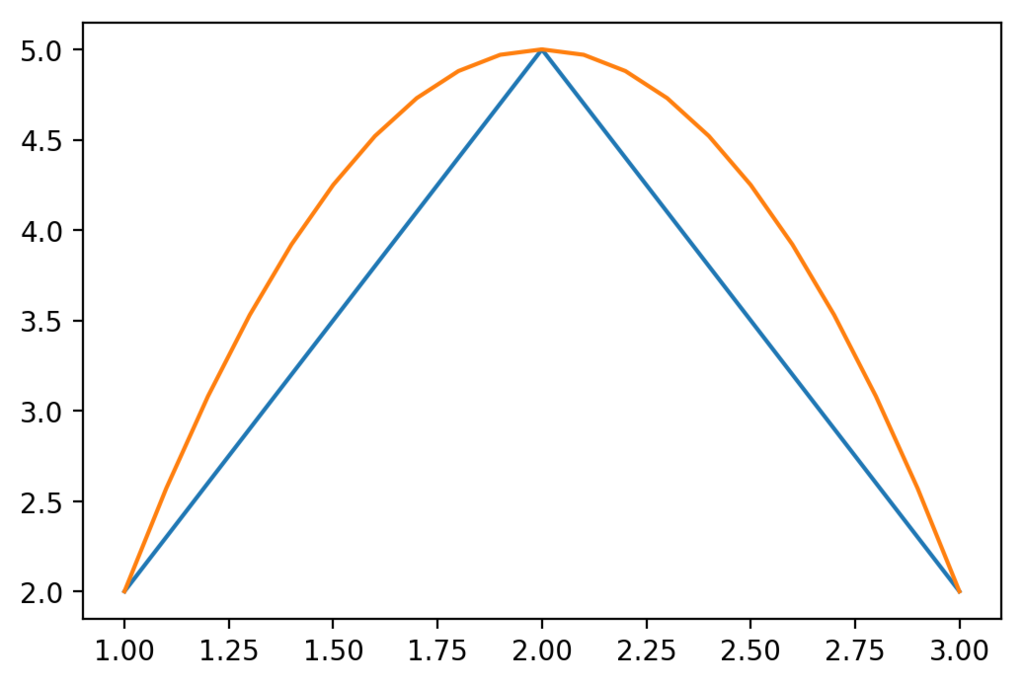
fit = pylab.polyfit([1, 2, 3], [2, 5, 2], 2) predictedDistances = pylab.polyval(fit, [1, 2, 3]) pylab.plot([1, 2, 3], predictedDistances) arange = pylab.arange(1, 3.1, 0.1) predictedDistances = pylab.polyval(fit, arange) pylab.plot(arange, predictedDistances)
[<matplotlib.lines.Line2D at 0x23e1d58a2b0>]
18.2 発射体の振舞い
code 18.4
メモ
8.3.4. defaultdict オブジェクトの練習で少し書き直した。
また、僕が入手したlauncherData.txtは区切り文字がスペースとタブが入り乱れているアホみたいなファイルだったので、スペース区切りに直した。あと、ついでにcsv.Snifferの使い方を練習した。csv.Snifferは、入力されたファイルに対する適切なDialectのサブクラスを返す。
def getTrajectoryData(fileName): data = collections.defaultdict(list) with open(fileName, 'rt', newline='') as fin: dialect = csv.Sniffer().sniff(fin.read(2048)) # csv.Snifferの練習 fin.seek(0) # sniff関数内でfinでの位置が先頭からずれてしまっている(今回は終端まで来てしまっている)ため、先頭に戻さないといけない。さもなくばgetTrajectoryDataは意図しないデータ(空のリスト等)を返す。 csvReader = csv.reader(fin, dialect) for line in csvReader: try: # ヘッダーはハンドリングする。浮動小数に変換することができないヘッダーをfloat関数に渡したときにValueErrorがでることを利用した。 for i in range(len(line)): data[i].append(float(line[i])) except ValueError: continue return (data[0], [data[i] for i in data][1:]) def processTrajectories(fileName): distances, heights = getTrajectoryData(fileName) numTrials = len(heights) distances = pylab.array(distances) totHeights = pylab.array(len(distances)*[0.0]) for h in heights: totHeights += pylab.array(h) meanHeights = totHeights/numTrials # テキストでは、numTrialsを計算していたのを忘れていたのか?使わせてもらった。 pylab.title(f'Trajectory of Projectile (Mean of {numTrials} Trials)') pylab.xlabel('Inches from Launch Point') pylab.ylabel('Inches Above Launch Point') pylab.plot(distances, meanHeights, 'ko') fit = pylab.polyfit(distances, meanHeights, 1) altitudes = pylab.polyval(fit, distances) pylab.plot(distances, altitudes, 'b', label='Linear Fit') fit = pylab.polyfit(distances, meanHeights, 2) altitudes = pylab.polyval(fit, distances) # テキストはミスだろうな。なんで2回もpolyfitやってんねんって感じ。 pylab.plot(distances, altitudes, 'b', label='Quadratic Fit') pylab.legend()
launcherDataTxt = 'launcherData.txt'
メモ
なぜか、横軸は等間隔でとられておらず、1000付近の方が観測地点が多いようだ。
processTrajectories(launcherDataTxt)

18.2.1 決定係数
code 18.5
def rSquared(measured, predicted): estimateError = ((predicted - measured)**2).sum() meanOfMeasured = stat.mean(measured) variability = ((measured - meanOfMeasured)**2).sum() return 1 - estimateError/variability
def getTrajectoryData(fileName): data = collections.defaultdict(list) with open(fileName, 'rt', newline='') as fin: dialect = csv.Sniffer().sniff(fin.read(2048)) # csv.Snifferの練習 fin.seek(0) # sviff関数内でfinでの位置が先頭からずれてしまっている(今回は終端まで来てしまっている)ため、先頭に戻さないといけない。さもなくばgetTrajectoryDataは意図しないデータ(空のリスト等)を返す。 csvReader = csv.reader(fin, dialect) for line in csvReader: try: # ヘッダーはハンドリングする。浮動小数に変換することができないヘッダーをfloat関数に渡したときにValueErrorがでることを利用した。 for i in range(len(line)): data[i].append(float(line[i])) except ValueError: continue return (data[0], [data[i] for i in data][1:]) def processTrajectories(fileName): distances, heights = getTrajectoryData(fileName) numTrials = len(heights) distances = pylab.array(distances) totHeights = pylab.array(len(distances)*[0.0]) for h in heights: totHeights += pylab.array(h) meanHeights = totHeights/numTrials # テキストでは、numTrialsを計算していたのを忘れていたのか?使わせてもらった。 pylab.title(f'Trajectory of Projectile (Mean of {numTrials} Trials)') pylab.xlabel('Inches from Launch Point') pylab.ylabel('Inches Above Launch Point') pylab.plot(distances, meanHeights, 'ko') fit = pylab.polyfit(distances, meanHeights, 1) altitudes = pylab.polyval(fit, distances) pylab.plot(distances, altitudes, 'b', label='Linear Fit') print(f'RSquare of linear fit = {rSquared(meanHeights, altitudes)}') fit = pylab.polyfit(distances, meanHeights, 2) altitudes = pylab.polyval(fit, distances) # テキストはミスだろうな。なんで2回もpolyfitやってんねんって感じ。 pylab.plot(distances, altitudes, 'b', label='Quadratic Fit') print(f'RSquare of quadratic fit = {rSquared(meanHeights, altitudes)}') pylab.legend()
processTrajectories(launcherDataTxt)
RSquare of linear fit = 0.0177433205440769
RSquare of quadratic fit = 0.9857653692869693
18.2.2 計算モデルを使う
code 18.6
def getHorizontalSpeed(quadfit, minX, maxX): inchesPerFoot = 12 xMid = (maxX - minX)/2 a, b, c = quadfit[0], quadfit[1], quadfit[2] yPeak = a*xMid**2 + b*xMid + c g = 32.16*inchesPerFoot t = (2*yPeak/g)**0.5 print(f'Horizontal speed = {int(xMid/(t*inchesPerFoot))}feet/sec')
def getTrajectoryData(fileName): data = collections.defaultdict(list) with open(fileName, 'rt', newline='') as fin: dialect = csv.Sniffer().sniff(fin.read(2048)) # csv.Snifferの練習 fin.seek(0) # sviff関数内でfinでの位置が先頭からずれてしまっている(今回は終端まで来てしまっている)ため、先頭に戻さないといけない。さもなくばgetTrajectoryDataは意図しないデータ(空のリスト等)を返す。 csvReader = csv.reader(fin, dialect) for line in csvReader: try: # ヘッダーはハンドリングする。浮動小数に変換することができないヘッダーをfloat関数に渡したときにValueErrorがでることを利用した。 for i in range(len(line)): data[i].append(float(line[i])) except ValueError: continue return (data[0], [data[i] for i in data][1:]) def processTrajectories(fileName): distances, heights = getTrajectoryData(fileName) numTrials = len(heights) distances = pylab.array(distances) totHeights = pylab.array(len(distances)*[0.0]) for h in heights: totHeights += pylab.array(h) meanHeights = totHeights/numTrials # テキストでは、numTrialsを計算していたのを忘れていたのか?使わせてもらった。 pylab.title(f'Trajectory of Projectile (Mean of {numTrials} Trials)') pylab.xlabel('Inches from Launch Point') pylab.ylabel('Inches Above Launch Point') pylab.plot(distances, meanHeights, 'ko') fit = pylab.polyfit(distances, meanHeights, 1) altitudes = pylab.polyval(fit, distances) pylab.plot(distances, altitudes, 'b', label='Linear Fit') fit = pylab.polyfit(distances, meanHeights, 2) altitudes = pylab.polyval(fit, distances) # テキストはミスだろうな。なんで2回もpolyfitやってんねんって感じ。 pylab.plot(distances, altitudes, 'b', label='Quadratic Fit') pylab.legend() getHorizontalSpeed(fit, distances[-1], distances[0]) # テキストは絶対ミスやろなぁ・・・getHorizontalSpeedの第一引数は二次多項式の係数のはずだからこれが正解。
processTrajectories(launcherDataTxt)
Horizontal speed = 136feet/sec
18.3 指数的に分布するデータに適合させる
code 18.7
def plotFittingPowerOf3(stop=10): xVals = pylab.arange(stop) # テキストではfor文を使って3**iの数値から成るリストを作成していたが、arrayを使った方が簡単だろう。 vals = 3**xVals # 2行で終わる。 pylab.plot(vals, 'ko', label='Actual points') fit = pylab.polyfit(xVals, vals, 5) yVals = pylab.polyval(fit, xVals) pylab.plot(yVals, 'kx', label='Predicted points', markeredgewidth=2, markersize=15) # markersize=25はでかすぎるので15にしておいた。 pylab.title('Fitting y = 3**x') pylab.legend(loc = 'upper left')
ちなみに、図 18.9のタイトルが2**xになっているが、3**xの間違いだろう。
plotFittingPowerOf3()

def plotFittingPowerOf3(stop=10): xVals = pylab.arange(stop) # テキストではfor文を使って3**iの数値から成るリストを作成していたが、arrayを使った方が簡単だろう。 vals = 3**xVals # 2行で終わる。 pylab.plot(vals, 'ko', label='Actual points') fit = pylab.polyfit(xVals, vals, 5) yVals = pylab.polyval(fit, xVals) pylab.plot(yVals, 'kx', label='Predicted points', markeredgewidth=2, markersize=15) # markersize=25はでかすぎるので15にしておいた。 pylab.title('Fitting y = 3**x') pylab.legend(loc = 'upper left') print(f'Model predicts that 3**20 is roughly {pylab.polyval(fit, [3**20][0])}') print(f'Actual value of 3**20 = {3**20}')
plotFittingPowerOf3()
Model predicts that 3**20 is roughly 2.4547827637212492e+48
Actual value of 3**20 = 3486784401
図 18.10のプロット
def plotFig1810(stop=10): vals = 3**pylab.arange(stop) pylab.plot(vals, 'ko', label='Actual points') pylab.semilogy()
plotFig1810()

code 18.8
def createData(f, xVals): # この関数作る必要あるのか? return f(xVals)
def fitExpData(xVals, yVals): logVals = math.log(yVals, 2) fit = pylab.polyfit(xVals, logVals, 1) return fit, 2.0
def plotFittingExpFunc(stop=10, anyX=20): xVals = pylab.arange(stop) f = lambda x: 3**x yVals = createData(f, xVals) pylab.plot(xVals, yVals, 'ko', label='Actual values') fit, base = fitExpData(xVals, yVals) predictedYVals = base**(pylab.polyval(fit, xVals)) pylab.plot(xVals, predictedYVals, label = 'Predicted values') pylab.title('Fitting an Exponential Function') pylab.legend(loc='upper left') print(f'f({anyX}) = {f(anyX)}') print(f'Predicted value = {int(base**(pylab.polyval(fit, [anyX])))}') # テキストのylabってなんだよw
メモ
今書いたplotFittingExpFunc関数は以下のエラーが出る。原因はfitExpData関数の中のmath.log(yVals, 2)にある。[Python]exp,sin等の関数に引数として配列を渡したい時を参考にして解決した。
plotFittingExpFunc()
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
<ipython-input-189-bc73174cc2e3> in <module>
----> 1 plotFittingExpFunc()
<ipython-input-188-16a8181357ef> in plotFittingExpFunc(stop, anyX)
4 yVals = createData(f, xVals)
5 pylab.plot(xVals, yVals, 'ko', label='Actual values')
----> 6 fit, base = fitExpData(xVals, yVals)
7 predictedYVals = base**(pylab.polyval(fit, xVals))
8 pylab.plot(xVals, predictedYVals, label = 'Predicted values')
<ipython-input-187-40ad0566ff29> in fitExpData(xVals, yVals)
1 def fitExpData(xVals, yVals):
----> 2 logVals = math.log(yVals, 2)
3 fit = pylab.polyfit(xVals, logVals, 1)
4 return fit, 2.0
TypeError: only size-1 arrays can be converted to Python scalars

以下のfitExpData関数を使えばエラー無く実行できる。
def fitExpData(xVals, yVals): # logVals = math.log(yVals, 2) logVals = pylab.log2(yVals) fit = pylab.polyfit(xVals, logVals, 1) return fit, 2.0
plotFittingExpFunc()
f(20) = 3486784401
Predicted value = 3486784401

最後に
この本読んでるけど、コードが冗長なとこが結構あるな。僕の性格的にこういうのがおおいと結構げんなりする。けど、同時にある程度奇麗に書き直せた時は快感だから引き続き続けていこう。

世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
- 作者: ジョン・V.グッターグ,John V. Guttag,久保幹雄
- 出版社/メーカー: 近代科学社
- 発売日: 2017/09/01
- メディア: 単行本
- この商品を含むブログを見る
Python言語によるプログラミングイントロダクション 第2版 写経(17章)
世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
の写経をしたので記録を公開しておきます。 この記事では、17章をまとめておきます。
Jupyter Labで実行したものをペーストしているので、ちょっとした正誤表になるかもしれません。
以下、使用モジュールとpylabの設定。
import pylab import statistics as stat pylab.rc('figure', dpi=200) import random import scipy as sp import scipy.integrate import scipy.stats
写経
17.1 ボストンマラソンの標本抽出
openとreadlineは
7. 入力と出力
を参照した。
csvモジュール使った方が絶対簡単だけど、別の方法のファイル操作を勉強するためにも今回はテキスト通りにやる。ただ、withは使う。
code 17.2
def getBMData(filename): data = {'name': [], 'gender': [], 'age':[], 'division': [], 'country': [], 'time': []} with open(filename) as f: for line in f.readlines(): split = line.split(',') data['name'].append(split[0]) data['gender'].append(split[1]) data['age'].append(split[2]) data['division'].append(split[3]) data['country'].append(split[4]) data['time'].append(float(split[5][:-1])) return data def makeHist(data, bins, title, xLabel, yLabel): pylab.hist(data, bins) pylab.title(title) pylab.xlabel(xLabel) pylab.ylabel(yLabel) mean = stat.mean(data) std = stat.stdev(data) pylab.annotate('Mean = ' + str(round(mean, 2)) + '\nSD = ' + str(round(std, 2)), fontsize=20, xy=(0.65, 0.75), xycoords = 'axes fraction')
bmResults2012Txt='bm_results2012.txt' times = getBMData(bmResults2012Txt)['time']
makeHist(times, 20, '2012 Boston Marathon', 'Minutes to Complete Race', 'Number of Runners')

code 17.3
def sampleTimes(times, numExamples): sample = random.sample(times, numExamples) makeHist(sample, 10, 'Sample of Size ' + str(numExamples), 'Minutes to Complete Race', 'Number of Runners')
sampleSize = 40
sampleTimes(times, sampleSize)

code 17.4
area = round(scipy.integrate.quad(sp.stats.norm.pdf, -3, 3, (0, 1))[0], 4) print('Probability of being within 3', 'of true of tight dist. =', area) area = round(scipy.integrate.quad(sp.stats.norm.pdf, -3, 3, (0, 100))[0], 4) print('Probability of being within 3', 'of true of tight dist. =', area)
Probability of being within 3 of true of tight dist. = 0.9973
Probability of being within 3 of true of tight dist. = 0.0239
code 17.5
def testSamples(numTrials, sampleSize): tightMeans, wideMeans = [], [] for t in range(numTrials): sampleTight, sampleWide = [], [] for i in range(sampleSize): sampleTight.append(random.gauss(0, 1)) sampleWide.append(random.gauss(0, 100)) tightMeans.append(stat.mean(sampleTight)) wideMeans.append(stat.mean(sampleWide)) return tightMeans, wideMeans tightMeans, wideMeans = testSamples(1000, 40) pylab.plot(wideMeans, 'y*', label=' SD = 100') pylab.plot(tightMeans, 'bo', label='SD = 1') pylab.xlabel('Sample Number') pylab.ylabel('Sample Mean') pylab.title('Means of Samples of Size ' + str(40)) pylab.legend() pylab.figure() pylab.hist(wideMeans, bins=20, label='SD = 100') pylab.title('Distribution of Sample Means') pylab.xlabel('Sample Mean') pylab.ylabel('Frequency of Occurrence') pylab.legend()
<matplotlib.legend.Legend at 0x17d790c4c18>


17.2 中心極限定理
code 17.6
def plotMeans(numDicePerTrial, numDiceThrown, numBins, legend, color, style): means = [] numTrials = numDiceThrown//numDicePerTrial for i in range(numTrials): vals = 0 for j in range(numDicePerTrial): vals += 5*random.random() means.append(vals/numDicePerTrial) pylab.hist(means, numBins, color=color, label=legend, weights=pylab.array(len(means)*[1])/len(means), hatch=style) return stat.mean(means), stat.variance(means) mean, var = plotMeans(1, 100000, 11, '1 die', 'w', '*') print('Mean of rolling 1 die =', round(var, 4), 'Variance =', round(var, 4)) mean, var = plotMeans(100, 100000, 11, 'Mean of 100 die', 'w', '//') print('Mean of rolling 100 die =', round(var, 4), 'Variance =', round(var, 4)) pylab.title('Rolling Continuous Dice') pylab.xlabel('Value') pylab.ylabel('Probability') pylab.legend()
Mean of rolling 1 die = 2.0828 Variance = 2.0828
Mean of rolling 100 die = 0.0215 Variance = 0.0215
<matplotlib.legend.Legend at 0x215d97df7b8>

メモ
weights=pylab.array(len(means)*[1])/len(means)はlen(means)*[1/len(means)]でもできる。前者は、numpy.ndarrayオブジェクトに含まれる要素すべてに対して演算(/len(means))を一度に実行させることで長さがlen(mean)で、各要素が1/len(means)のnumpy.ndarrayオブジェクトを生成している。ただ、同じことは後者のlen(means)*[1/len(means)]でもできるしわかりやすい。
以下はnumpy.ndarrayオブジェクトに割り算をしたメモ。その下のコードはlen(means)*[1/len(means)]を使用したバージョン。
array = pylab.array([1, 2, 3])
type(array)
numpy.ndarray
array/3
array([0.33333333, 0.66666667, 1. ])
means = [2, 2, 3, 2, 3, 3, 4, 2, 1]
len(means)*[1/len(means)]
[0.1111111111111111,
0.1111111111111111,
0.1111111111111111,
0.1111111111111111,
0.1111111111111111,
0.1111111111111111,
0.1111111111111111,
0.1111111111111111,
0.1111111111111111]
def plotMeans(numDicePerTrial, numDiceThrown, numBins, legend, color, style): means = [] numTrials = numDiceThrown//numDicePerTrial for i in range(numTrials): vals = 0 for j in range(numDicePerTrial): vals += 5*random.random() means.append(vals/numDicePerTrial) pylab.hist(means, numBins, color=color, label=legend, weights=len(means)*[1/len(means)], hatch=style) return stat.mean(means), stat.variance(means) mean, var = plotMeans(1, 100000, 11, '1 die', 'w', '*') print('Mean of rolling 1 die =', round(var, 4), 'Variance =', round(var, 4)) mean, var = plotMeans(100, 100000, 11, 'Mean of 100 die', 'w', '//') print('Mean of rolling 100 die =', round(var, 4), 'Variance =', round(var, 4)) pylab.title('Rolling Continuous Dice') pylab.xlabel('Value') pylab.ylabel('Probability') pylab.legend()
Mean of rolling 1 die = 2.0798 Variance = 2.0798
Mean of rolling 100 die = 0.0208 Variance = 0.0208
<matplotlib.legend.Legend at 0x215d9e76b38>

weightsはpylab.histの第一引数と同じ形のアレイ(今のところ長さが同じリストっぽいものというイメージ)を指定する。weightsの各値はpylab.histの第一引数の各値に重みづけをする。
pylab.hist([1, 1, 2], weights=[10, 0, 3])
(array([10., 0., 0., 0., 0., 0., 0., 0., 0., 3.]),
array([1. , 1.1, 1.2, 1.3, 1.4, 1.5, 1.6, 1.7, 1.8, 1.9, 2. ]),
<a list of 10 Patch objects>)

どうやら、weightsには負の値も指定できるようだ。
pylab.hist([1, 1, 2], weights=[10, -12, 3])
(array([-2., 0., 0., 0., 0., 0., 0., 0., 0., 3.]),
array([1. , 1.1, 1.2, 1.3, 1.4, 1.5, 1.6, 1.7, 1.8, 1.9, 2. ]),
<a list of 10 Patch objects>)

code 17.7
def plotEstOfMeanFinTime(minimum=50, maximum=2000, interval=200, numPerSampleSize=20): meanOfMeans, stdOfMeans = [], [] sampleSizes = range(minimum, maximum, interval) for sampleSize in sampleSizes: sampleMeans = [] for t in range(numPerSampleSize): sample = random.sample(times, sampleSize) sampleMeans.append(stat.mean(sample)) meanOfMeans.append(stat.mean(sampleMeans)) stdOfMeans.append(stat.stdev(sampleMeans)) pylab.errorbar(sampleSizes, meanOfMeans, yerr=1.96*pylab.array(stdOfMeans), label='Estimated mean and 95% confidence interval') pylab.xlim(0, max(sampleSizes) + 50) pylab.axhline(stat.mean(times), linestyle='--', label='Population mean') pylab.title('Estimates of Mean Finishing Time') pylab.xlabel('Sample Size') pylab.ylabel('Finishing Time (minutes)') pylab.legend(loc='best')
plotEstOfMeanFinTime(numPerSampleSize=20)

各sampleSize毎に200回サンプリングしてみたら、やはり分散は安定して減少する。
plotEstOfMeanFinTime(numPerSampleSize=200)

メモ
テキストの図 17.6をプロットしてみた。
def oneOverSquare(x): return 1/(x)**0.5 popStd = stat.pstdev(times) def plotSEvsSD(minimum=1, maximum=2000, interval=50, numPerSampleSize=20): stdOfMeans = [] sampleSizes = range(minimum, maximum, interval) for sampleSize in sampleSizes: sampleMeans = [] for t in range(numPerSampleSize): sample = random.sample(times, sampleSize) sampleMeans.append(stat.mean(sample)) stdOfMeans.append(stat.stdev(sampleMeans)) pylab.plot(sampleSizes, stdOfMeans, label=f'Std of {numPerSampleSize} Means') pylab.plot(range(minimum, maximum), popStd*oneOverSquare(pylab.array([i for i in range(minimum, maximum)])), linestyle='--', label='SEM') pylab.xlim(min(sampleSizes), max(sampleSizes) + interval) pylab.title(f'SE vs. SD of {numPerSampleSize} Means') pylab.xlabel('Sample Size') pylab.ylabel('Standard Deviation') pylab.legend(loc='best')
plotSEvsSD()

sampleSizeが大きくなると、サンプリング平均の標準偏差がSEMを下回る傾向があるんだけど、これは何なんだろう?統計に自信ニキ教えてください。
plotSEvsSD(minimum=2000, maximum=20000, interval=1000, numPerSampleSize=100)

17.3 平均の標準誤差
code 17.8
def plotSamSDvsPopSD(start=2, stop=200, step=2, numPerSample=100): popStd = stat.stdev(times) sampleSizes = range(start, stop, step) diffsMeans = [] for sampleSize in sampleSizes: diffs = [] for t in range(numPerSample): diffs.append(abs(popStd - stat.stdev(random.sample(times, sampleSize)))) diffsMeans.append(stat.mean(diffs)) pylab.plot(sampleSizes, diffsMeans) pylab.xlabel('Sample Size') pylab.ylabel('Abs(Pop. Std - Sample Std)') pylab.title('Sample SD vs Population SD')
plotSamSDvsPopSD()

code 17.9
def extOutsideConfInterval(sampleSize=200, numTrial=10000): popMean = stat.mean(times) numBad = 0 for t in range(numTrial): sample = random.sample(times, sampleSize) sampleMean = stat.mean(sample) se = stat.stdev(sample)/sampleSize**0.5 if abs(popMean - sampleMean) > 1.96*se: numBad += 1 print(f'Fraction outside 95% confidence interval = {numBad/numTrial}')
extOutsideConfInterval()
Fraction outside 95% confidence interval = 0.0544

世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
- 作者: ジョン・V.グッターグ,John V. Guttag,久保幹雄
- 出版社/メーカー: 近代科学社
- 発売日: 2017/09/01
- メディア: 単行本
- この商品を含むブログを見る
Python言語によるプログラミングイントロダクション 第2版 写経(16章)
世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
の写経をしたので記録を公開しておきます。 この記事では、16章をまとめておきます。
Jupyter Labで実行したものをペーストしているので、ちょっとした正誤表になるかもしれません。
以下、使用モジュール。
import random import statistics as stat import pylab pylab.rc('figure', dpi=400)
写経
code 16.1
def rollDie(): return random.choice([1, 2, 3, 4, 5, 6]) def checkPascal(numTrials): numWins = 0 for i in range(numTrials): for j in range(24): d1 = rollDie() d2 = rollDie() if d1 == 6 and d2 == 6: numWins += 1 break print('Probability of winning = ', numWins/numTrials)
checkPascal(1000000)
Probability of winning = 0.49219
code 16.2
class CrapsGame(object): def __init__(self): self.passWins, self.passLosses = 0, 0 self.dpWins, self.dpLosses, self.dpPushes = 0, 0, 0 def playHand(self): throw = rollDie() + rollDie() if throw == 7 or throw == 11: self.passWins += 1 self.dpLosses += 1 elif throw == 2 or throw == 3 or throw == 12: self.passLosses += 1 if throw == 12: self.dpPushes += 1 else: self.dpWins += 1 else: point = throw while True: throw = rollDie() + rollDie() if throw == point: self.passWins += 1 self.dpLosses += 1 break elif throw == 7: self.passLosses += 1 self.dpWins += 1 break def passResults(self): return (self.passWins, self.passLosses) def dpResults(self): return (self.dpWins, self.dpLosses, self.dpPushes)
code 16.3
def crapSim(handsPerGame, numGames): games = [] for t in range(numGames): c = CrapsGame() for i in range(handsPerGame): c.playHand() games.append(c) pROIPerGames, dpROIPerGames = [], [] for g in games: wins, losses = g.passResults() pROIPerGames.append((wins - losses)/float(handsPerGame)) wins, losses, pushes = g.dpResults() dpROIPerGames.append((wins - losses)/float(handsPerGame)) meanROI = str(round(100*stat.mean(pROIPerGames)/numGames, 4)) + '%' sigma = str(round(100*stat.stdev(pROIPerGames), 4)) + '%' print('Pass:', 'Mean ROI =', meanROI, 'Std. Dev. =', sigma) meanROI = str(round(100*stat.mean(dpROIPerGames)/numGames, 4)) + '%' sigma = str(round(100*stat.stdev(dpROIPerGames), 4)) + '%' print('Don\'t pass:', 'Mean ROI =', meanROI, 'Std. Dev. =', sigma)
crapSim(20, 20)
Pass: Mean ROI = 0.15% Std. Dev. = 18.0933%
Don't pass: Mean ROI = -0.3125% Std. Dev. = 19.3904%
crapSim(1000000, 10)
Pass: Mean ROI = -0.1492% Std. Dev. = 0.1136%
Don't pass: Mean ROI = -0.1286% Std. Dev. = 0.1083%
crapSim(20, 1000000)
Pass: Mean ROI = -0.0% Std. Dev. = 22.3581%
Don't pass: Mean ROI = -0.0% Std. Dev. = 22.044%
code 16.4
class CrapsGame(object): def __init__(self): self.passWins, self.passLosses = 0, 0 self.dpWins, self.dpLosses, self.dpPushes = 0, 0, 0 def playHand(self): pointsDict = {4: 1/3, 5: 2/5, 6: 5/11, 8: 5/11, 9: 2/5, 10: 1/3} throw = rollDie() + rollDie() if throw == 7 or throw == 11: self.passWins += 1 self.dpLosses += 1 elif throw == 2 or throw == 3 or throw == 12: self.passLosses += 1 if throw == 12: self.dpPushes += 1 else: self.dpWins += 1 else: if random.random() <= pointsDict[throw]: self.passWins += 1 self.dpLosses += 1 else: self.passLosses += 1 self.dpWins += 1 def passResults(self): return (self.passWins, self.passLosses) def dpResults(self): return (self.dpWins, self.dpLosses, self.dpPushes)
def rollDie(): return random.choice([1, 1, 2, 3, 3, 4, 4, 5, 5, 5, 6, 6])
crapSim(1000000, 10)
Pass: Mean ROI = 0.2299% Std. Dev. = 0.0927%
Don't pass: Mean ROI = -0.508% Std. Dev. = 0.0978%
code 16.5
def throwNeedles(numNeedles): inCircle = 0 for Needles in range(numNeedles): # テキストだと、1, 1+numNeedlesって書いてあったけど、for文の中でNeedles変数を使用するところなさそうだし、これでいいはず。 x = random.random() y = random.random() if (x*x + y*y)**0.5 <= 1: inCircle += 1 return 4*(inCircle/float(numNeedles)) def getEst(numNeedles, numTrials): estimate = [] for t in range(numTrials): piGuess = throwNeedles(numNeedles) estimate.append(piGuess) sDev = stat.stdev(estimate) curEst = stat.mean(estimate) print('Est. =' + str(round(curEst, 5)) + ',', 'Std. dev. = ' + str(round(sDev, 5)) + ',', 'Needles = ' + str(numNeedles)) # テキストはprint関数の括弧が閉じてない気がする。 return (curEst, sDev) def estPi(precision, numTrials): numNeedles = 1000 sDev = precision while sDev >= precision/1.96: curEst, sDev = getEst(numNeedles, numTrials) numNeedles *= 4 # じれったいので4倍で増やすことにした return curEst
estPi(0.01, 100)
Est. =3.13016, Std. dev. = 0.05046, Needles = 1000
Est. =3.14358, Std. dev. = 0.03472, Needles = 2000
Est. =3.14173, Std. dev. = 0.02282, Needles = 4000
Est. =3.14166, Std. dev. = 0.01758, Needles = 8000
Est. =3.14183, Std. dev. = 0.01209, Needles = 16000
Est. =3.14296, Std. dev. = 0.00865, Needles = 32000
Est. =3.14094, Std. dev. = 0.0062, Needles = 64000
Est. =3.14217, Std. dev. = 0.00492, Needles = 128000
3.142170625
curEst ± precisionの線をプロットしたヒストグラムを書いてみた。 確かに、平均の近くに95%以上のデータが含まれていそうだということが確認できた。
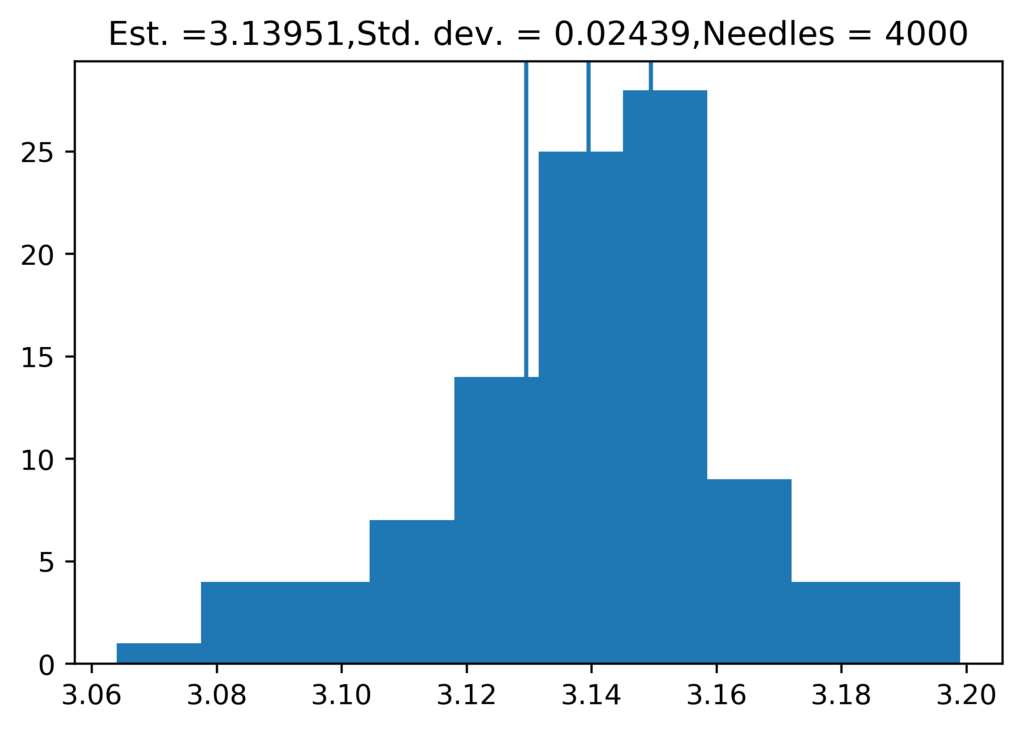
def throwNeedles(numNeedles): inCircle = 0 for Needles in range(numNeedles): # テキストだと、1, 1+numNeedlesって書いてあったけど、for文の中でNeedles変数を使用するところなさそうだし、これでいいはず。 x = random.random() y = random.random() if (x*x + y*y)**0.5 <= 1: inCircle += 1 return 4*(inCircle/float(numNeedles)) def getEst2(numNeedles, numTrials, precision): """precisionはestPi関数から参照することを前提としている""" estimate = [] for t in range(numTrials): piGuess = throwNeedles(numNeedles) estimate.append(piGuess) sDev = stat.stdev(estimate) curEst = stat.mean(estimate) title = 'Est. =' + str(round(curEst, 5)) + ',' + 'Std. dev. = ' + str(round(sDev, 5)) + ',' + 'Needles = ' + str(numNeedles) pylab.figure() pylab.title(title) pylab.hist(estimate) pylab.axvline(curEst - precision) pylab.axvline(curEst) pylab.axvline(curEst + precision) return (curEst, sDev) def estPi(precision, numTrials): numNeedles = 1000 sDev = precision while sDev >= precision/1.96: curEst, sDev = getEst2(numNeedles, numTrials, precision=precision) numNeedles *= 4 # じれったいので16倍で増やすことにした return curEst
estPi(0.01, 100)
3.142018125




疑問
Markdownでlatexを使用するのが成功しなかった・・・ Markdownはやめた方が良いのか?どなたかMarkdownでlatexを書く方法をご存知の方がいましたら教えてください。

世界標準MIT教科書 Python言語によるプログラミングイントロダクション第2版: データサイエンスとアプリケーション
- 作者: ジョン・V.グッターグ,John V. Guttag,久保幹雄
- 出版社/メーカー: 近代科学社
- 発売日: 2017/09/01
- メディア: 単行本
- この商品を含むブログを見る
Pythonのcsvモジュールで出力されるファイルの改行コードをLFにする
目的
Pythonの標準モジュールであるcsvモジュールで出力されるファイルの改行コードの変更方法をまとめます。
方法
以下のようにして改行コードを指定します。writerの引数にlineterminator='\n'を指定すれば改行コードをLFに指定することができます。
with open('test.csv', 'at', newline='') as cout: writer = csv.writer(cout, lineterminator='\n') # lineterminatorを'\n'で上書きし、改行コードがLFになるようにした。 writer.writerow([2, 2, 2])
試しに、何も指定しない以下のコードも同時に実行して結果を見てみます。実際に改行コードの変更が確認できます。
with open('test.csv', 'at', newline='') as cout: writer = csv.writer(cout) writer.writerow([1, 1, 1])

サクラエディタでの改行コードの表示がCRLFとLFの二つになっているということが確認できます。
csvモジュールが使用する改行コードはデフォルトではCRLFを改行コードに使用することになっています。デフォルトのまま出力したファイルをLinux環境などで使用すると予期しないエラーに遭遇することもある(僕が昨日そうでした)ので、改行コードの指定は明示的に行っておいてもよいかもしれません。

- 作者: Bill Lubanovic,斎藤康毅,長尾高弘
- 出版社/メーカー: オライリージャパン
- 発売日: 2015/12/01
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (3件) を見る

- 作者: 柴田淳
- 出版社/メーカー: SBクリエイティブ
- 発売日: 2016/12/22
- メディア: 単行本
- この商品を含むブログ (2件) を見る



